PCR 聚合酶
PCR 优化的一般指导原则
概述
NEB 针对基于 PCR 的应用提供多种 DNA 聚合酶。在产品的说明书或产品的网页介绍上可以找到针对不同产品的特定优化建议。下面的一般指导原则将帮助您的
PCR 实验顺利进行。
反应建立指南
DNA 模板
• 尽可能使用高质量、经过纯化的
DNA 模板。扩增未纯化的DNA 时(菌落 PCR 或直接
PCR),请参阅特定 的产品信息
• 对于低复杂度的模板(如质粒、λ或BACDNA),每
50 μl 反应体系加入 1 pg-10
ng DNA
• 对于高复杂度的模板(如基因组DNA),每 50 μl 反应体系加入
1 ng-1 μg DNA
• DNA 浓度增高会降低扩增特异性,尤其是循环次数较多时
引物
• 一般长度为 20-40 个核苷酸
• 理想的 GC 含量为 40-60%
• 引物的 Tm 值可通过 NEB 的
Tm Calculator 计算 (www.neb.com/TmCalculator)
• 退火温度应根据特定酶的推荐温度设定。请注意
Q5® 和 Phusion®*
推荐的退火温度是特别的
• 两条引物间 Tm 差值应在 5℃ 以内
• 避免引物形成引物内二级结构(如发卡结构)以及引物二聚体
• 每条引物的终浓度应为 0.05-1 μM。请参阅特定酶的详细推荐信息
• 引物浓度过高可能导致非特异性引物结合,并产生非特异性扩增产物
• 当扩增产物的大小超过 20 kb 时,引物长度应≥ 24 个核苷酸,GC 含量应在
50% 以上,Tm 值应在 60℃ 以上
• 当引入限制性位点时,需要在识别位点
5´ 端额外添加 6 个碱基
• 为了消除引物降解以及后续的非特异扩增,可使用热启动聚合酶(如
One Taq® 热启动 DNA聚合酶或
Q5 热启动超保真 DNA 聚合酶)
镁离子浓度
• 对于大多数 PCR 聚合酶,Mg2+ 最佳浓度通常为 1.5-2.0 mM
• NEB 提供的大多数 PCR 缓冲液在 1 倍浓度下已含有足够浓度的
Mg2+关于 Mg2+ 请参阅特定酶的产品信息
• NEB 提供多种不含 Mg2+ 的缓冲液,在需要严格控制
Mg2+ 浓度的实验中,可向其中补加 Mg2+
• 如果需要,可通过将 Mg2+ 浓度增加 0.2–1 mM来进一步优化。在某些特定试验中,聚合酶可能需要高达6mM 的镁离子
• Mg2+ 浓度过高可能导致出现非预期
PCR 产物
• Mg2+ 浓度过低可能导致反应失败
dNTPs
• 理想的 dNTP 浓度通常为每种 200 μM,但是某些聚合酶可能需要每种 dNTP 的浓度高达 400 μM。请参阅特定产品的说明书获取详细的推荐信息
• 过量的 dNTP 会螯合 Mg2+ 并抑制聚合酶的活性
• dNTP 浓度过低可增加保真性,但会减少产量
• 当使用古生菌 PCR 聚合酶时,引物、模板或dNTP 混合液中的尿嘧啶会导致反应失败。针对这类反应可使OneTaq 或 Taq DNA 聚合酶聚合酶浓度
• 反应中的最佳聚合酶浓度因酶而异。请参阅特定产品的说明书获取详细的推荐信息
• 一般而言,过量的聚合酶会导致扩增失败,特别是扩增长片段时
建立反应
• 除非使用热启动聚合酶(如 OneTaq 热启动DNA
聚合酶或 Q5 热启动超保真 DNA 聚合酶)否则需在冰上混合各种反应组份
• 尽可能最后加入聚合酶
• 建立好反应体系后立即放入已经预热至变性温度的热循环仪中。请注意在使用热启动聚合酶(如
OneTaq 热启动 DNA 聚合酶或Q5
热启动超保真 DNA 聚合酶)时无需预热热循环仪
循环条件指南
变性
• 最佳的变性温度范围为 94℃-98℃,并因酶而异。请参阅特定产品的说明书获取详细的推荐信息
• 避免长时间或高温度温育(除非模板富含
GC)
• 通常,热循环过程中变性时间为
5-30 秒
• NEB 提供基于核酸适配子的热启动聚合酶,无需额外变性步骤以激活聚合酶
退火
• 引物的 Tm 值可通过 NEB 的
Tm Calculator 计算 (www.neb.com/TmCalculator)
• 除 Q5 超保真 DNA 聚合酶或
Phusion 超保真DNA 聚合酶*以外,退火温度一般设定为比引物对的最低
Tm 值低 2℃-5℃
• 使用 Q5 超保真 DNA 聚合酶或
Phusion 超保真 DNA 聚合酶*时,退火温度一般设定为比引物对的最低
Tm 值高 0℃-3℃。请参阅特定产品的参考文献获取详细的推荐信息
• 通过优化退火温度或改用热启动
DNA 聚合酶(如 OneTaq 热启动
DNA 聚合酶或 Q5 热启动超保真 DNA 聚合酶)可避免形成非特异产物
• 通过温度梯度 PCR 可优化退火温度,将起始温度设定为比引物对的最低 Tm 值低 5℃
• 理想状态下,引物 Tm 值应比延伸温度低。但是,如果计算出的 Tm 值比延伸温度高,可采用两步法 PCR(将退火和延伸合并为一步)
延伸
• 推荐的延伸反应温度范围为65℃-72℃,因酶而异。请参阅特定产品的说明书获取详细的推荐信息
• 延伸速率因酶而异。一般而言,延伸速率范围为15-60
s/kb。请参阅特定产品的说明书获取详细的推荐信息
• 过长的延伸时间会增加错配率,增加非特异扩增和/或减少扩增产量
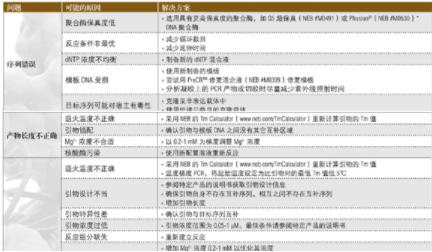
PCR 问题解决指南
下面的指南可用于解决 PCR 反应中出现的问题。关于优化反应的更多建议可访问 NEB 网站的技术参考部分。
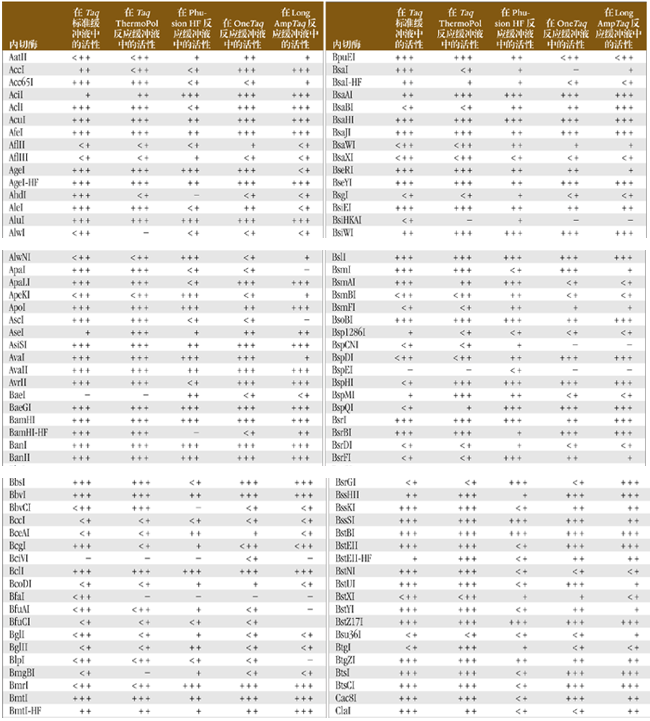
内切酶在 PCR 混合物中的活性
通常,PCR 反应后还要对产物进行酶切才能进行下一步工作。为了方便起见,可以将内切酶直接加到 PCR 反应产物中,而省略纯化 PCR 产物的步骤。下表归纳了在 Taq、Phusion*、OneTaq 和 LongAmpTaq PCR 产物混合物中各种限制性内切酶的酶切活性。50 μl 反应体系中,将 5 单位内切酶加入到 PCR 反应产物中,在适当的温度下反应 1 小时,凝胶电泳分析酶活性。PCR 反应产物中包括:1X ThermoPol 反应缓冲液、标准 Taq 反应缓冲液、Phusion HF 反应缓冲液、OneTaq 标准反应缓冲液或 LongAmpTaq 反应缓冲液,含 dNTPs(终浓度为 200 μM)、1 μg DNA 和 1 单位 Taq DNA 聚合酶。
注意:聚合酶在切割 DNA 后仍然有活性,并能改变切割的 DNA 片段末端,对后续的连接反应造成影响。含有限制性内切酶识别位点的引物在酶消化过程中有竞争抑制作用。在非最佳条件下进行酶切反应容易出现星号活性。如果遇到上述任何问题,则需用乙醇沉淀,酚/氯仿抽提或离心柱纯化 DNA。
图例
使用 5 单位内切酶切割 PCR 混合物:
+++ 表示完全切割
++ 表示 ~ 50% 被切割
+ 表示 ~ 25% 被切割 -
表示未被切割
北京百灵克生物科技有限责任公司
公司地址:北京海淀区永定路88号长银大厦9C05
咨询热线:010-58895880/58896088
助力科研 托举未来
- 上一篇:蛋白表达 2020/3/23
- 下一篇:复旦交大牵头开发新冠病毒mRNA疫苗取得实质性进展! 2020/3/20
